Laboratorio del corso di Biochimica del Metabolismo
Corso di Laurea in Biotecnologie
Produzione di una Proteina Ricombinante
Il
sistema di espressione
In termini generali, per far esprimere una proteina in un batterio, abbiamo bisogno di una molecola di DNA circolare (chiamata vettore o plasmide) che possa essere efficacemente replicata all'interno della cellula ospite. La molecola di DNA deve essere circolare come il genoma del batterio, per non essere degradata dai meccanismi di difesa del batterio contro il DNA estraneo, e contenere una "origine di replicazione" simile a quella del suo ospite per poter essere duplicata. Perchè questa molecola di DNA venga mantenuta efficacemente all'interno della cellula ospite, questa deve conferire un vantaggio ai batteri che la acquisiscono: in genere la capacità di sopravvivere in presenza di un antibiotico altrimenti nocivo per l'ospite. Vogliamo anche che all'interno di ciascuna cellula vi siano più copie del plasmide, al fine di aumentare il numero di copie del gene di interesse che deve essere espresso e anche quelle del gene che conferisce resistenza antibiotica alle cellule che contengono il plasmide. Il vettore contiene una sequenza con funzione di promotore (in grado cioè di reclutare la RNA polimerasi e permettere la trascrizione della sequenza di DNA che lo segue) e una codificante un segnale di riconoscimento per i ribosomi (responsabili della traduzione dell'RNA messaggero in proteine), sufficienti a garantire la trascrizione e la traduzione del gene di interesse localizzato
a valle. I sistemi utilizzati in genere sono ancora un pochino più sofisticati.
Informazioni utili
Innanzi tutto, nella gran parte dei casi, è utile ritardare l'espressione per permettere ai batteri di crescere adeguatamente prima di iniziare a produrre la proteina "ospite" (e questo è particolarmente importante nei casi in cui la produzione della proteina possa avere effetti tossici); vogliamo in oltre fare in modo che il livello di espressione sia quantitativamente abbondante, anche per facilitarre i successivi passaggi di purificazione. Questo tipo di considerazioni hanno favorito lo sviluppo di nuovi vettori e la parallela modifica dei loro ospiti.
Una delle evoluzioni più comuni è rappresentata in questo schema:
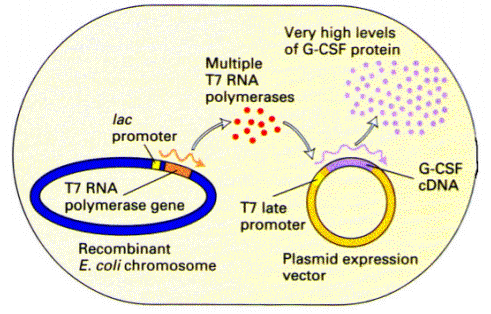
| Fig. 1. Sistema di espressione a due stadi, basato sull'impiego della RNA polimerasi del fago T7 e di uno dei suoi promotori tardivi. Il gene codificante la RNA polimerasi virale, sotto il controllo di un promotore inducibile, è stato stabilmente integrato nel genoma di un ceppo di E. coli specificamente ingegnerizzato. Il gene di interesse è inserito in un plasmide a valle di uno dei promotori più forti del fago T7 (che però non è riconoscibile dalla polimerasi batterica). La sintesi della RNA polimerasi fagica (a seguito dell'aggiunta di IPTG) permette la trascrizione del gene di interesse che è preceduto da un promotore virale. Il plasmide rappresentato nella figura è solo una delle molte copie presenti in ogni cellula. (Fonte: in MCB IV, fig. 7.37) |
Questo sistema sfrutta le potenzialità di alcuni elementi virali. Nel plasmide utilizzato come vettore di espressione è infatti presente un promotore proveniente dal fago T7(chiamiamo fago un virus che infetta i batteri). Si tratta di un promotore molto efficiente che, tuttavia, per funzionare ha bisogno della RNA polimerasi virale, non essendo riconosciuto dall'analoga polimerasi batterica. Per questo motivo, utilizzando le tecniche dell'ingegneria genetica, nel genoma batterico è stato integrato il gene codificante l'enzima virale. L'espressione di questo gene è però a sua volta controllata e può essere attivata solo dall'aggiunta di IPTG.
Solo per i più curiosi di voi la trascrizione della RNAsi fagica è controllata da una sequenza derivata dal promotore dell'operone lattosio. Questo sito e normalmente occupato dal repressore dell'operone lattosio, che è sempre presente nella cellula e impedisce la trascrizione del gene che controlla. Soltanto l'aggiunta di IPTG sarà in grado di rimuovere il repressore e permettere la sintesi della polimerasi fagica. Conseguentemente, sarà quindi attivata la trascrizione del gene di interesse a partire dal promotore virale presente nel plasmide. Questo sistema, quindi, soddisfa due requisiti fondamentali: l'inducibilità, che permette di superare eventuali problemi legati alla tossicità della proteina, e l'efficienza dell'espressione tipica dei sistemi virali. Il vettore e il batterio ospite utilizzati nel nostro esperimento riflettono sostanzialmente la filosofia di questo approccio.
Il
vettore: pET28a+
Come si può notare dalla mappa del plasmide :
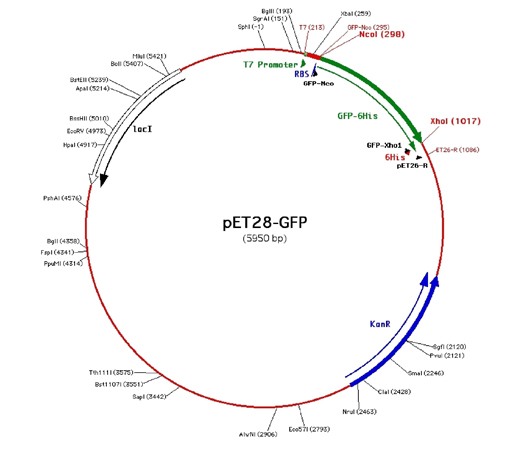
|
questo vettore possiede, in aggiunta al promotore virale, il sito di legame per i ribosomi ed un terminatore della trascrizione, un altro interessante elemento: il segmento di DNA che che si trova a valle della nostra SYFP, codificante un peptide costituito da 6 istidine. Tenendo conto del loro modulo di lettura, è quindi possibile produrre una SYFP (in figura indicata in modo generico come GFP) contenente, fusa all'estremità C-terminale, la coda di istidine. Proprio questo elemento, come vedremo, può facilitare enormemente la purificazione ed inoltre, dopo l'isolamento, ci consente di seguire una proteina qualsiasi (nel nostro caso la SYFP) utilizzando anticorpi commerciali specifici per la codina, chimata in gergo "tag". In molti casi, anche se non nel nostro, questo ed altri "tag" possono essere eliminati grazie alla presenza di un sito riconoscibile da una particolare proteasi.
L'ospite:
BL21(DE3)pLysS
Il ceppo batterico utilizzato in questa esercitazione, indicato con la sigla
BL21(DE3), fa parte di una famiglia piuttosto ampia di possibili ospiti
per vettori basati sull'utilizzo del promotore di T7 come appena descritto.
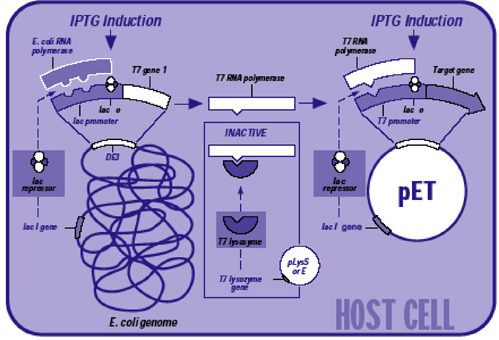
| Fig. 3. Schema delle cellule BL21(DE3)pLys alle quali appartiene il ceppo batterico utilizzato come ospite del plasmide pET28a+ |
Come si può vedere nella figura, queste cellule sono in grado di esprimere l'RNA polimerasi di un fago perchè nel loro genoma è stata integrata una sequenza (indicata con DE3) che contiene il gene dell'enzima. Come è già stato detto, l'espressione è inducibile perchè la regione promotoriale, essendo derivata dall'operone lattosio, è sotto controllo del repressore prodotto costitutivamente (ovvero sempre) dal gene del repressore chiamato lac I (presente sia nell'ospite che nel vettore) . Una ulteriore, interessante caratteristica del ceppo BL21 e dei suoi derivati è l'assenza di due proteasi (lon e ompT), una situazione che riduce la possibilità di degradazione delle proteine sintetizzate.